Ribonuclease H
| ribonuclease H | |||||||
|---|---|---|---|---|---|---|---|
| Estrutura cristalográfica de E. coli RNase HI.[1] | |||||||
| Indicadores | |||||||
| Número EC | 3.1.26.4 | ||||||
| Bases de dados | |||||||
| IntEnz | IntEnz | ||||||
| BRENDA | BRENDA | ||||||
| ExPASy | NiceZyme | ||||||
| KEGG | KEGG | ||||||
| MetaCyc | via metabólica | ||||||
| PRIAM | PRIAM | ||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||
| |||||||
| Ribonuclease retroviral H | |||||||
|---|---|---|---|---|---|---|---|
| Indicadores | |||||||
| Número EC | 3.1.26.13 | ||||||
| Bases de dados | |||||||
| IntEnz | IntEnz | ||||||
| BRENDA | BRENDA | ||||||
| ExPASy | NiceZyme | ||||||
| KEGG | KEGG | ||||||
| MetaCyc | via metabólica | ||||||
| PRIAM | PRIAM | ||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||
| |||||||
A ribonuclease H (RNase H ou RNH abreviada) é uma família de enzimas de endonuclease não específicas da sequência que catalisa a clivagem do RNA em um substrato de RNA/DNA por meio de um mecanismo hidrolítico. Os membros da família RNase H podem ser encontrados em quase todos os organismos, de bactérias a arquéias e eucariotos.
A família é dividida em grupos evolutivamente relacionados com preferências de substrato ligeiramente diferentes, ribonuclease H1 e H2 amplamente designada.[2] O genoma humano codifica H1 e H2. A ribonuclease humana H2 é um complexo heterotrimérico composto por três subunidades, mutações em qualquer uma das causas genéticas de uma doença rara conhecida como síndrome de Aicardi-Goutières.[3] Um terceiro tipo, intimamente relacionado ao H2, é encontrado apenas em alguns procariontes,[4] enquanto H1 e H2 ocorrem em todos os domínios da vida.[4] Adicionalmente, os domínios de ribonuclease H retroviral tipo RNase H1 ocorrem em proteínas de transcriptase reversa de múltiplos domínios, que são codificadas por retrovírus como o HIV e são necessárias para replicação viral.[5][6]
Nos eucariotos, a ribonuclease H1 está envolvida na replicação do DNA do genoma mitocondrial. Tanto o H1 quanto o H2 estão envolvidos em tarefas de manutenção do genoma, como o processamento de estruturas de R-loop.[2][7]
Classificação e nomenclatura
[editar | editar código-fonte]A ribonuclease H é uma família de enzimas de endonuclease com uma especificidade de substrato compartilhada para a cadeia de RNA dos duplexes de RNA-DNA. Por definição, as RNases H clivam o fosfodiéster do esqueleto de RNA para deixar um grupo 3'hidroxila e 5'fosfato.[7] As RNases H foram propostas como membros de uma superfamília evolutivamente relacionada que engloba outras nucleases e enzimas de processamento de ácidos nucléicos, tais como integrase retroviral, transposases de DNA, resolvases da junção de Holliday, proteínas Piwi e Argonaute, várias exonucleases e a proteína spliceossômica Prp8.[8][9]
As RNases H podem ser amplamente divididas em dois subtipos, H1 e H2, que por razões históricas recebem designações de números arábicos em eucariotos e designações de números romanos em procariotas. Assim, a Escherichia coli RNase HI é um homólogo do Homo sapiens RNase H1.[2][7] Em E. coli e em muitos outros procariotos, o gene rnhA codifica HI e o gene rnhB codifica HII. Uma terceira classe relacionada, chamada HIII, ocorre em algumas bactérias e arquéias; está intimamente relacionado às enzimas HII procarióticas.[4]
Estrutura
[editar | editar código-fonte]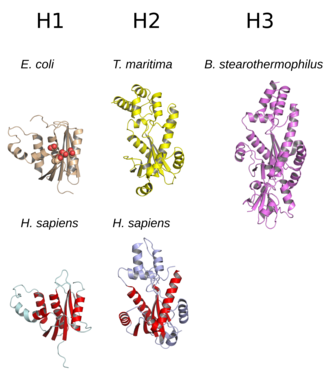
A estrutura da RNase H geralmente consiste em uma folha β de 5 filas cercada por uma distribuição de hélices α.[10] Todas as RNases H têm um sítio ativo centrado em um motivo de sequência conservada composto por resíduos de aspartato e glutamato, frequentemente chamado de motivo DEDD. Esses resíduos interagem com os íons de magnésio necessários cataliticamente.[7][5]
As RNases H2 são maiores que o H1 e geralmente têm hélices adicionais. A organização do domínio das enzimas varia; alguns membros procarióticos e a maioria dos eucarióticos do grupo H1 têm um pequeno domínio adicional no terminal N conhecido como "domínio de ligação híbrido", o que facilita a ligação aos duplexes híbridos RNA: DNA e às vezes confere maior processamento.[2][7][11] Embora todos os membros do grupo H1 e os membros procarióticos do grupo H2 funcionem como monômeros, as enzimas eucarióticas H2 são heterotrímeros obrigatórios.[2][7] As enzimas procarióticas HIII são membros do grupo H2 mais amplo e compartilham a maioria das características estruturais com H2, com a adição de um domínio de ligação à caixa TATA do terminal N.[7] Os domínios da RNase retroviral H que ocorrem nas proteínas da transcriptase reversa de múltiplos domínios têm estruturas muito semelhantes ao grupo H1.[5]
As RNases H1 foram extensivamente estudadas para explorar as relações entre estrutura e atividade enzimática. Eles também são usados, especialmente o homólogo de E. coli, como sistemas modelo para estudar a dobragem de proteínas.[12][13][14] Dentro do grupo H1, foi identificada uma relação entre maior afinidade de ligação ao substrato e a presença de elementos estruturais que consistem em uma hélice e alça flexível, proporcionando uma superfície maior e mais básica de ligação ao substrato. A hélice C possui uma distribuição taxonômica dispersa; está presente nos homólogos de E. coli e da RNase H1 humana e ausente no domínio HIV RNase H, mas existem exemplos de domínios retrovirais com hélices C.[15][16]
Função
[editar | editar código-fonte]As enzimas da ribonuclease H clivam as ligações fosfodiéster do RNA em um híbrido de RNA: DNA de fita dupla, deixando um grupo hidroxila 3' e um grupo fosfato 5' em cada extremidade do local de corte. A RNase H1 e H2 têm preferências distintas de substrato e funções distintas, mas sobrepostas, na célula. Nos procariontes e nos eucariontes inferiores, nenhuma enzima é essencial, ao passo que ambos são considerados essenciais nos eucariontes superiores.[2] A atividade combinada das enzimas H1 e H2 está associada à manutenção da estabilidade do genoma devido à degradação das enzimas do componente de RNA das alças R.[17][18]
Ribonuclease H1
[editar | editar código-fonte]| RNase H | |
|---|---|
| Indicadores | |
| Pfam | PF00075 |
| InterPro | IPR002156 |
| PROSITE | PS50879 |
As enzimas da ribonuclease H1 requerem pelo menos quatro pares de bases contendo ribonucleotídeo em um substrato e não podem remover um único ribonucleotídeo de uma fita que é de outra forma composta por desoxirribonucleotídeos. Por esse motivo, considera-se improvável que as enzimas RNase H1 estejam envolvidas no processamento de iniciadores de RNA de fragmentos de Okazaki durante a replicação do DNA.[2] A RNase H1 não é essencial em organismos unicelulares onde foi investigada; em E. coli, os nocautes da RNase H1 conferem um fenótipo sensível à temperatura,[7] e em S. cerevisiae, eles produzem defeitos na resposta ao estresse.[19]
Em muitos eucariotos, incluindo mamíferos, os genes da RNase H1 incluem uma sequência de direcionamento mitocondrial, levando à expressão de isoformas com e sem o MTS presente. Como resultado, a RNase H1 está localizada nas mitocôndrias e no núcleo. Nos modelos de camundongos knockout, os mutantes nulos de RNase H1 são letais durante a embriogênese devido a defeitos na replicação do DNA mitocondrial.[2][20][21] Os defeitos na replicação do DNA mitocondrial induzidos pela perda da RNase H1 provavelmente são devidos a defeitos no processamento do loop R.[18]
Ribonuclease H2
[editar | editar código-fonte]| RNase HII | |
|---|---|
| Indicadores | |
| Pfam | PF01351 |
| InterPro | IPR024567 |
Nos procariontes, a RNase H2 é enzimaticamente ativa como uma proteína monomérica. Nos eucariotos, é um heterotrímero obrigatório composto por uma subunidade catalítica A e subunidades estruturais B e C. Enquanto a subunidade A é intimamente homóloga à RNase H2 procariótica, as subunidades B e C não têm homólogos aparentes nos procariotas e são mal conservadas em o nível de sequência mesmo entre eucariotos.[22][23] A subunidade B medeia as interações proteína-proteína entre o complexo H2 e o PCNA, que localiza H2 nos focos de replicação.[24]
As enzimas H2 procarióticas e eucarióticas podem clivar ribonucleotídeos únicos em uma fita.[2] no entanto, eles têm padrões de clivagem e preferências de substrato ligeiramente diferentes: as enzimas procarióticas têm menor processabilidade e hidrolisam os ribonucleotídeos sucessivos com mais eficiência do que os ribonucleotídeos com um desoxirribonucleotídeo 5', enquanto as enzimas eucarióticas são mais processuais e hidrolisam os dois tipos de substrato com eficiência semelhante.[2][25] A especificidade do substrato da RNase H2 confere um papel no reparo por excisão de ribonucleotídeos, removendo ribonucleotídeos não incorporados do DNA, além do processamento da alça R.[26][27][24] Embora H1 e H2 estejam presentes no núcleo das células dos mamíferos, H2 é a fonte dominante da atividade da RNase H e é importante para manter a estabilidade do genoma.[24]
Alguns procariontes possuem um gene adicional do tipo H2 designado RNase HIII na nomenclatura do número romano usada para os genes procarióticos. As proteínas HIII estão mais intimamente relacionadas ao grupo H2 por identidade de sequência e semelhança estrutural, mas possuem preferências de substrato que se assemelham mais a H1.[7][28] Ao contrário de HI e HII, ambos amplamente distribuídos entre procariontes, o HIII é encontrado em apenas alguns organismos com distribuição taxonômica dispersa; é um pouco mais comum nas arqueias e raramente ou nunca é encontrado no mesmo genoma procariótico que o HI.[29]
Mecanismo
[editar | editar código-fonte]O local ativo de quase todas as RNases H contém quatro resíduos de aminoácidos carregados negativamente, conhecido como motivo DEDD; frequentemente histidina também está presente.[2][7]
Os resíduos carregados ligam um ou dois íons metálicos necessários para a catálise; sob condições fisiológicas, esses são íons magnésio, mas o manganês também geralmente suporta a atividade enzimática,[2][7] enquanto o cálcio pode inibi-lo.[11][30] Embora os mecanismos catalíticos de dois íons metálicos sejam muito comuns em enzimas envolvidas na bioquímica do fosfato, tem sido objeto de debate na literatura se um ou dois íons são utilizados na catálise da RNase H. Em qualquer mecanismo proposto, pelo menos uma molécula de água participa da reação.[31][32]
A maioria das evidências experimentais para o mecanismo de catálise da RNase H vem de medições realizadas em membros do grupo H1, geralmente o homólogo de E. coli . De acordo com as medições dessa proteína, um dos resíduos de aspartato tem um pKa elevado, enquanto outro tem um pKa anormalmente baixo.[33] Não está claro se algum dos resíduos do local ativo participa da reação como base geral.[7] Além disso, é possível que um dos átomos de oxigênio do substrato participe diretamente da reação como base.[34]
Na biologia humana
[editar | editar código-fonte]O genoma humano contém quatro genes que codificam a RNase H:
- RNASEH1, um exemplo do subtipo H1 (monomérico)
- RNASEH2A, a subunidade catalítica do complexo H2 trimérico
- RNASEH2B, uma subunidade estrutural do complexo H2 trimérico
- RNASEH2C, uma subunidade estrutural do complexo H2 trimérico
Além disso, o material genético de origem retroviral aparece frequentemente no genoma, refletindo a integração dos genomas dos retrovírus endógenos humanos. Tais eventos de integração resultam na presença de genes que codificam a transcriptase reversa retroviral, que inclui um domínio da RNase H. Um exemplo é o ERVK6.[35] Retrotransposons de repetição terminal longos também são comuns no genoma e geralmente incluem seus próprios domínios de RNase H, com uma história evolutiva complexa.[36][37]
Papel na doença
[editar | editar código-fonte]
Em pequenos estudos, mutações na RNase H1 humana foram associadas à oftalmoplegia externa progressiva crônica, uma característica comum da doença mitocondrial.[21]
Mutações em qualquer uma das três subunidades da RNase H2 estão bem estabelecidas como causas de um distúrbio genético raro conhecido como síndrome de Aicardi-Goutières (AGS),[3] que se manifesta como sintomas neurológicos e dermatológicos desde tenra idade.[39] Os sintomas da SAG se assemelham aos da infecção viral congênita e estão associados à regulação positiva inadequada do interferon tipo I. A AGS também pode ser causada por mutações em outros genes: TREX1, SAMHD1, ADAR e MDA5/IFIH1, todos envolvidos no processamento de ácidos nucleicos.[40] A caracterização da distribuição mutacional em uma população de pacientes com SGA encontrou 5% de todas as mutações com SGA no RNASEH2A, 36% em 2B e 12% em 2C.[41] As mutações em 2B foram associadas a um comprometimento neurológico um pouco mais leve[42] e a uma ausência de regulação positiva de genes induzida por interferon que pode ser detectada em pacientes com outros genótipos associados à SGA.[40]
Em vírus
[editar | editar código-fonte]
Dois grupos de vírus usam a transcrição reversa como parte de seus ciclos de vida: retrovírus, que codificam seus genomas em RNA de fita simples e se replicam por meio de um DNA intermediário de fita dupla; e vírus dsDNA-RT, que replicam seus genomas de DNA de fita dupla através de um intermediário de RNA "pré-genoma". Exemplos patogênicos incluem vírus da imunodeficiência humana e vírus da hepatite B, respectivamente. Ambos codificam proteínas grandes de transcriptase reversa multifuncional (RT) contendo domínios de RNase H.[44][45]
As proteínas retrovirais de RT do HIV-1 e o vírus da leucemia murina são os membros mais bem estudados da família.[46][47] O RT retroviral é responsável pela conversão do genoma de RNA de fita simples do vírus em DNA de fita dupla. Esse processo requer três etapas: primeiro, a a atividade da polimerase de DNA dependente de RNA produz DNA de fita negativa a partir do modelo de RNA de fita positiva, gerando um intermediário híbrido RNA: DNA; segundo, a cadeia de RNA é destruída; e terceiro, a atividade da polimerase de DNA dependente de DNA sintetiza o DNA de fita positiva, gerando DNA de fita dupla como produto final. A segunda etapa deste processo é realizada por um domínio RNase H localizado no terminal C da proteína RT.[5][6][48][49]
RNase H executa três tipos de acções: clivagem de degradação não-específica do genoma de ARN de cadeia mais, remoção específica do minus-strand tRNA iniciador, e remoção do iniciador além de cadeia rica em purinas do tracto polipurino (PPT).[50] A RNase H desempenha um papel na iniciação da cadeia positiva, mas não no método convencional de sintetizar uma nova sequência de iniciador. Em vez disso, a RNase H cria um "primer" do PPT que é resistente à clivagem da RNase H. Ao remover todas as bases, exceto o PPT, o PPT é usado como um marcador para o final da região U3 de sua repetição terminal longa.[49]
Como a atividade da RNase H é necessária para a proliferação viral, esse domínio foi considerado um alvo de drogas para o desenvolvimento de drogas antirretrovirais usadas no tratamento do HIV/AIDS e outras condições causadas por retrovírus. Foram identificados inibidores da RNase retroviral H de vários quimiotipos diferentes, muitos dos quais possuem um mecanismo de ação baseado na quelação dos cátions do sítio ativo.[51] Os inibidores da transcriptase reversa que inibem especificamente a função da polimerase da RT estão em uso clínico generalizado, mas não são inibidores da função da RNase H; é a única função enzimática codificada pelo HIV que ainda não é alvo de medicamentos em uso clínico.[48][52]
Evolução
[editar | editar código-fonte]As RNases H são amplamente distribuídas e ocorrem em todos os domínios da vida. A família pertence a uma superfamília maior de enzimas nuclease[8][9] e é considerada evolutivamente antiga.[53] Nos genomas procarióticos, múltiplos genes da RNase H estão frequentemente presentes, mas há pouca correlação entre a ocorrência dos genes HI, HII e HIII e as relações filogenéticas gerais, sugerindo que a transferência horizontal de genes pode ter desempenhado um papel no estabelecimento da distribuição dessas enzimas. A RNase HI e HIII raramente ou nunca aparecem no mesmo genoma procariótico. Quando o genoma de um organismo contém mais de um gene da RNase H, eles às vezes apresentam diferenças significativas no nível de atividade. Essas observações foram sugeridas para refletir um padrão evolutivo que minimiza a redundância funcional entre os genes da RNase H.[7][29] A RNase HIII, que é exclusiva dos procariontes, tem uma distribuição taxonômica dispersa e é encontrada tanto nas bactérias quanto nas arqueias;[29] acredita-se que tenha divergido do HII bastante cedo.[54]
A trajetória evolutiva da RNase H2 em eucariotos, especialmente o mecanismo pelo qual os homólogos eucarióticos se tornaram heterotrímeros obrigatórios, não é clara; as subunidades B e C não possuem homólogos aparentes em procariontes.[2][23]
Formulários
[editar | editar código-fonte]Como a RNase H especificamente degrada apenas o RNA em híbridos de RNA: DNA de fita dupla, é comumente usada como reagente de laboratório em biologia molecular . Preparações purificadas de E. coli RNase HI e HII estão disponíveis comercialmente. A RNase HI é frequentemente usada para destruir o molde de RNA após a síntese complementar de DNA (cDNA) da primeira fita por transcrição reversa. Também pode ser usado para clivar sequências específicas de RNA na presença de pequenos segmentos complementares de DNA.[55] Técnicas altamente sensíveis, como ressonância plasmônica de superfície, podem ser usadas para detecção.[56][57] A RNase HII pode ser usada para degradar o componente iniciador de RNA de um fragmento de Okazaki ou para introduzir cortes de fita simples em posições contendo um ribonucleotídeo.[55] Uma variante da PCR de arranque a quente, conhecida como PCR dependente de RNase H ou rhPCR, foi descrita usando uma RNase HII termoestável do arco hipertermofílico Pyrococcus abyssi.[58] De notar, a proteína inibidora da ribonuclease normalmente utilizada como reagente não é eficaz na inibição da atividade de HI ou HII.[55]
História
[editar | editar código-fonte]As ribonucleases H foram descobertas no laboratório de Peter Hausen quando os pesquisadores descobriram a atividade da endonuclease híbrida RNA: DNA no timo de bezerros em 1969 e deram o nome de "ribonuclease H " para designar sua especificidade híbrida.[22][59][60] A atividade da RNase H foi posteriormente descoberta em E. coli[61] e em uma amostra de oncovírus com genomas de RNA durante os primeiros estudos de transcrição reversa viral.[62][63] Mais tarde ficou claro que o extrato de timo de bezerro continha mais de uma proteína com atividade da RNase H[64] e que E. coli continha dois genes da RNase H.[65][66] Originalmente, a enzima agora conhecida como RNase H2 nos eucariotos foi designada H1 e vice-versa, mas os nomes das enzimas eucarióticas foram trocados para coincidir com os de E. coli para facilitar a análise comparativa, produzindo a nomenclatura moderna na qual as enzimas procarióticas são designadas. com algarismos romanos e as enzimas eucarióticas com algarismos arábicos.[2][22][67][68] A RNase HIII procariótica, relatada em 1999, foi o último subtipo de RNase H a ser identificado.[67]
A caracterização da RNase H2 eucariótica foi historicamente um desafio, em parte devido à sua baixa abundância.[2] Esforços cuidadosos na purificação da enzima sugeriram que, ao contrário da E. coli RNase H2, a enzima eucariótica tinha várias subunidades.[69] O homólogo de S. cerevisiae da proteína E. coli (ou seja, a subunidade H2A) foi facilmente identificável pela bioinformática quando o genoma da levedura foi sequenciado,[70] mas a proteína correspondente foi encontrada sem atividade enzimática isolada.[2][19] Eventualmente, as subunidades B e C da levedura foram isoladas por co-purificação e consideradas necessárias para a atividade enzimática.[71] No entanto, as subunidades B e C da levedura têm uma identidade de sequência muito baixa para seus homólogos em outros organismos, e as proteínas humanas correspondentes foram identificadas conclusivamente somente após mutações nos três causarem a síndrome de Aicardi-Goutières.[2][3]
- ↑ PDB 1JL1; Goedken ER, Marqusee S (Dezembro de 2001). «Native-state energetics of a thermostabilized variant of ribonuclease HI». Journal of Molecular Biology. 314 (4): 863–71. PMID 11734003. doi:10.1006/jmbi.2001.5184
- ↑ a b c d e f g h i j k l m n o p q «Ribonuclease H: the enzymes in eukaryotes». The FEBS Journal. 276. PMC 2746905
. PMID 19228196. doi:10.1111/j.1742-4658.2009.06908.x
- ↑ a b c Crow YJ, Leitch A, Hayward BE, Garner A, Parmar R, Griffith E, et al. (August 2006). "Mutations in genes encoding ribonuclease H2 subunits cause Aicardi-Goutières syndrome and mimic congenital viral brain infection". Nature Genetics. 38 (8): 910–6. doi:10.1038/ng1842. PMID 16845400.
- ↑ a b c «Crystal structure of RNase H3-substrate complex reveals parallel evolution of RNA/DNA hybrid recognition». Nucleic Acids Research. 42. PMC 4132731
. PMID 25016521. doi:10.1093/nar/gku615
- ↑ a b c d «Crystal structure of the ribonuclease H domain of HIV-1 reverse transcriptase». Science. 252. PMID 1707186. doi:10.1126/science.1707186
- ↑ a b «Identification and characterization of HIV-specific RNase H by monoclonal antibody». The EMBO Journal. 7. PMC 454263
. PMID 2452083. doi:10.1002/j.1460-2075.1988.tb02805.x
- ↑ a b c d e f g h i j k l m «Ribonuclease H: molecular diversities, substrate binding domains, and catalytic mechanism of the prokaryotic enzymes». The FEBS Journal. 276. PMID 19228197. doi:10.1111/j.1742-4658.2009.06907.x
- ↑ a b «The RNase H-like superfamily: new members, comparative structural analysis and evolutionary classification». Nucleic Acids Research. 42. PMC 3985635
. PMID 24464998. doi:10.1093/nar/gkt1414
- ↑ a b «Retroviral integrases and their cousins». Current Opinion in Structural Biology. 6. ISSN 0959-440X. PMID 8696976. doi:10.1016/s0959-440x(96)80098-4
- ↑ «Thermal and mechanical multistate folding of ribonuclease H». The Journal of Chemical Physics. 131. PMID 20025349. doi:10.1063/1.3270167
- ↑ a b «Specific recognition of RNA/DNA hybrid and enhancement of human RNase H1 activity by HBD». The EMBO Journal. 27. PMC 2323259
. PMID 18337749. doi:10.1038/emboj.2008.44
- ↑ «Direct Observation of the Three-State Folding of a Single Protein Molecule». Science. 309. ISSN 0036-8075. PMID 16179479. doi:10.1126/science.1116702
- ↑ «A Thermodynamic Comparison of Mesophilic and Thermophilic Ribonucleases H». Biochemistry. 38. ISSN 0006-2960. PMID 10090773. doi:10.1021/bi982684h
- ↑ «The kinetic folding intermediate of ribonuclease H resembles the acid molten globule and partially unfolded molecules detected under native conditions». Nature Structural & Molecular Biology. 4. doi:10.1038/nsb0497-298
- ↑ «RNase H activity: structure, specificity, and function in reverse transcription». Virus Research. 134. PMC 2464458
. PMID 18261820. doi:10.1016/j.virusres.2007.12.007
- ↑ «Ribonuclease H: properties, substrate specificity and roles in retroviral reverse transcription». FEBS Journal. 276. ISSN 1742-4658. PMC 2742777
. PMID 19228195. doi:10.1111/j.1742-4658.2009.06909.x
- ↑ «RNase H enables efficient repair of R-loop induced DNA damage». eLife. 5. PMC 5215079
. PMID 27938663. doi:10.7554/eLife.20533
- ↑ a b «Viable RNaseH1 knockout mice show RNaseH1 is essential for R loop processing, mitochondrial and liver function». Nucleic Acids Research. 44. PMC 4914116
. PMID 27131367. doi:10.1093/nar/gkw350
- ↑ a b «The absence of ribonuclease H1 or H2 alters the sensitivity of Saccharomyces cerevisiae to hydroxyurea, caffeine and ethyl methanesulphonate: implications for roles of RNases H in DNA replication and repair». Genes to Cells. 5. PMID 11029655. doi:10.1046/j.1365-2443.2000.00373.x
- ↑ «Failure to produce mitochondrial DNA results in embryonic lethality in Rnaseh1 null mice». Molecular Cell. 11. PMID 12667461. doi:10.1016/s1097-2765(03)00088-1
- ↑ a b «RNASEH1 Mutations Impair mtDNA Replication and Cause Adult-Onset Mitochondrial Encephalomyopathy». American Journal of Human Genetics. 97. PMC 4572567
. PMID 26094573. doi:10.1016/j.ajhg.2015.05.013
- ↑ a b c Hollis T, Shaban NM (1 de janeiro de 2011). Nicholson, ed. Ribonucleases. Col: Nucleic Acids and Molecular Biology. [S.l.: s.n.] ISBN 9783642210778. doi:10.1007/978-3-642-21078-5_12
- ↑ a b «Contributions of the two accessory subunits, RNASEH2B and RNASEH2C, to the activity and properties of the human RNase H2 complex». Nucleic Acids Research. 37. PMC 2615623
. PMID 19015152. doi:10.1093/nar/gkn913
- ↑ a b c «Ribonuclease H2 in health and disease». Biochemical Society Transactions. 42. PMID 25109948. doi:10.1042/BST20140079
- ↑ «Contributions of the two accessory subunits, RNASEH2B and RNASEH2C, to the activity and properties of the human RNase H2 complex». Nucleic Acids Research. 37. PMC 2615623
. PMID 19015152. doi:10.1093/nar/gkn913
- ↑ «RNase H and multiple RNA biogenesis factors cooperate to prevent RNA:DNA hybrids from generating genome instability». Molecular Cell. 44. PMC 3271842
. PMID 22195970. doi:10.1016/j.molcel.2011.10.017
- ↑ «Mutagenic processing of ribonucleotides in DNA by yeast topoisomerase I». Science. 332. PMC 3380281
. PMID 21700875. doi:10.1126/science.1205016
- ↑ «Identification of the genes encoding Mn2+-dependent RNase HII and Mg2+-dependent RNase HIII from Bacillus subtilis: classification of RNases H into three families». Biochemistry. 38. PMID 9888800. doi:10.1021/bi982207z
- ↑ a b c «Evolution of ribonuclease H genes in prokaryotes to avoid inheritance of redundant genes». BMC Evolutionary Biology. 7. PMC 1950709
. PMID 17663799. doi:10.1186/1471-2148-7-128
- ↑ «Calcium inhibition of ribonuclease H1 two-metal ion catalysis». Journal of the American Chemical Society. 136. PMC 3985467
. PMID 24499076. doi:10.1021/ja411408x
- ↑ «Two-metal ion mechanism of RNA cleavage by HIV RNase H and mechanism-based design of selective HIV RNase H inhibitors». Nucleic Acids Research. 31. PMC 290251
. PMID 14627818. doi:10.1093/nar/gkg881
- ↑ «Making and breaking nucleic acids: two-Mg2+-ion catalysis and substrate specificity». Molecular Cell. 22. PMID 16600865. doi:10.1016/j.molcel.2006.03.013
- ↑ «Individual Ionization Constants of All the Carboxyl Groups in Ribonuclease HI from Escherichia coli Determined by NMR». Biochemistry. 33. ISSN 0006-2960. doi:10.1021/bi00183a034
- ↑ «Phosphodiester Cleavage in Ribonuclease H Occurs via an Associative Two-Metal-Aided Catalytic Mechanism». Journal of the American Chemical Society. 130. ISSN 0002-7863. PMC 2745632
. PMID 18662000. doi:10.1021/ja8005786
- ↑ «Genomic organization of the human endogenous retrovirus HERV-K(HML-2.HOM) (ERVK6) on chromosome 7». Genomics. 72. PMID 11401447. doi:10.1006/geno.2000.6488
- ↑ «Convergent evolution of ribonuclease h in LTR retrotransposons and retroviruses». Molecular Biology and Evolution. 32. PMC 4408406
. PMID 25605791. doi:10.1093/molbev/msv008
- ↑ «Ribonuclease H evolution in retrotransposable elements». Cytogenetic and Genome Research. 110. PMID 16093691. doi:10.1159/000084971
- ↑ «The structural and biochemical characterization of human RNase H2 complex reveals the molecular basis for substrate recognition and Aicardi-Goutières syndrome defects». The Journal of Biological Chemistry. 286. PMC 3060507
. PMID 21177858. doi:10.1074/jbc.M110.181974
- ↑ «Aicardi-Goutieres syndrome». British Medical Bulletin. 89. PMID 19129251. doi:10.1093/bmb/ldn049
- ↑ a b «Aicardi-Goutières syndrome and the type I interferonopathies». Nature Reviews. Immunology. 15. PMID 26052098. doi:10.1038/nri3850
- ↑ Crow YJ, Chase DS, Lowenstein Schmidt J, Szynkiewicz M, Forte GM, Gornall HL, et al. (February 2015). "Characterization of human disease phenotypes associated with mutations in TREX1, RNASEH2A, RNASEH2B, RNASEH2C, SAMHD1, ADAR, and IFIH1". American Journal of Medical Genetics. Part A. 167A (2): 296–312. doi:10.1002/ajmg.a.36887. PMC 4382202. PMID 25604658.
- ↑ Rice G, Patrick T, Parmar R, Taylor CF, Aeby A, Aicardi J, et al. (October 2007). "Clinical and molecular phenotype of Aicardi-Goutieres syndrome". American Journal of Human Genetics. 81 (4): 713–25. doi:10.1086/521373. PMC 2227922. PMID 17846997.
- ↑ «Crystal structure of HIV-1 reverse transcriptase in complex with a polypurine tract RNA:DNA». The EMBO Journal. 20. ISSN 0261-4189. PMC 145536
. PMID 11250910. doi:10.1093/emboj/20.6.1449
- ↑ «Molecular biology of hepatitis B virus infection». Virology. 479–480. PMC 4424072
. PMID 25759099. doi:10.1016/j.virol.2015.02.031
- ↑ Moelling, Karin; Broecker, Felix; Kerrigan, JohnE. (1 de janeiro de 2014). Vicenzi; Poli, eds. Human Retroviruses. Col: Methods in Molecular Biology. 1087. [S.l.: s.n.] ISBN 9781627036696. PMID 24158815. doi:10.1007/978-1-62703-670-2_7
- ↑ «Insight into the mechanism of the stabilization of moloney murine leukaemia virus reverse transcriptase by eliminating RNase H activity». Bioscience, Biotechnology, and Biochemistry. 74. PMID 20139597. doi:10.1271/bbb.90777
- ↑ «Murine leukemia virus reverse transcriptase: structural comparison with HIV-1 reverse transcriptase». Virus Research. 134. PMC 2443788
. PMID 18294720. doi:10.1016/j.virusres.2008.01.001
- ↑ a b Nowotny, Marcin; Figiel, Małgorzata (1 de janeiro de 2013). LeGrice; Gotte, eds. Human Immunodeficiency Virus Reverse Transcriptase. [S.l.: s.n.] ISBN 9781461472902. doi:10.1007/978-1-4614-7291-9_3
- ↑ a b «HIV-1 Ribonuclease H: Structure, Catalytic Mechanism and Inhibitors». Viruses. 2. PMC 3185654
. PMID 21994660. doi:10.3390/v2040900
- ↑ «Uncovering the complexities of retroviral ribonuclease H reveals its potential as a therapeutic target». AIDS Reviews. 4. PMID 12555693
- ↑ «HIV-1 RT-Associated RNase H Function Inhibitors: Recent Advances in Drug Development». Current Medicinal Chemistry. 17. doi:10.2174/092986710792065045
- ↑ «Recent progress in the research of small molecule HIV-1 RNase H inhibitors». Current Medicinal Chemistry. 21. ISSN 1875-533X. PMID 24438523. doi:10.2174/0929867321666140120121158
- ↑ «Characters of very ancient proteins». Biochemical and Biophysical Research Communications. 366. PMID 18073136. doi:10.1016/j.bbrc.2007.12.014
- ↑ «Evolutionary history of the TBP-domain superfamily». Nucleic Acids Research. 41. ISSN 0305-1048. PMC 3597702
. PMID 23376926. doi:10.1093/nar/gkt045
- ↑ a b c Nichols, Nicole M.; Yue, Dongxian (1 de janeiro de 2001). Current Protocols in Molecular Biology. Chapter 3. [S.l.: s.n.] ISBN 9780471142720. PMID 18972385. doi:10.1002/0471142727.mb0313s84
- ↑ «A non-PCR SPR platform using RNase H to detect MicroRNA 29a-3p from throat swabs of human subjects with influenza A virus H1N1 infection». The Analyst. 140. PMID 26000345. doi:10.1039/C5AN00679A
- ↑ «Direct detection of genomic DNA by enzymatically amplified SPR imaging measurements of RNA microarrays». Journal of the American Chemical Society. 126. PMID 15053580. doi:10.1021/ja039823p
- ↑ «RNase H-dependent PCR (rhPCR): improved specificity and single nucleotide polymorphism detection using blocked cleavable primers». BMC Biotechnology. 11. PMC 3224242
. PMID 21831278. doi:10.1186/1472-6750-11-80
- ↑ «Enzyme from Calf Thymus Degrading the RNA Moiety of DNA-RNA Hybrids: Effect on DNA-Dependent RNA Polymerase». Science. 166. ISSN 0036-8075. PMID 5812039. doi:10.1126/science.166.3903.393
- ↑ «Ribonuclease H». European Journal of Biochemistry. 14. ISSN 1432-1033. doi:10.1111/j.1432-1033.1970.tb00287.x
- ↑ «Ribonuclease H (hybrid) in Escherichia coli. Identification and characterization». The Journal of Biological Chemistry. 248. ISSN 0021-9258. PMID 4572736
- ↑ «Association of viral reverse transcriptase with an enzyme degrading the RNA moiety of RNA-DNA hybrids». Nature New Biology. 234. ISSN 0090-0028. PMID 4331605. doi:10.1038/newbio234240a0
- ↑ «Ribonuclease H: a ubiquitous activity in virions of ribonucleic acid tumor viruses». Journal of Virology. 10. ISSN 0022-538X. PMC 356594
. PMID 4118867
- ↑ «Distinct Ribonuclease H Activities in Calf Thymus». European Journal of Biochemistry. 52. ISSN 1432-1033. doi:10.1111/j.1432-1033.1975.tb03985.x
- ↑ «DNA sequence of the gene coding for Escherichia coli ribonuclease H». The Journal of Biological Chemistry. 258. ISSN 0021-9258. PMID 6296074
- ↑ «Isolation and characterization of a second RNase H (RNase HII) of Escherichia coli K-12 encoded by the rnhB gene». Proceedings of the National Academy of Sciences of the United States of America. 87. ISSN 0027-8424. PMC 55002
. PMID 2172991. doi:10.1073/pnas.87.21.8587
- ↑ a b «Identification of the genes encoding Mn2+-dependent RNase HII and Mg2+-dependent RNase HIII from Bacillus subtilis: classification of RNases H into three families». Biochemistry. 38. ISSN 0006-2960. PMID 9888800. doi:10.1021/bi982207z
- ↑ Crouch, R. J.; Arudchandran, A.; Cerritelli, S. M. (1 de janeiro de 2001). RNase H1 of Saccharomyces cerevisiae: methods and nomenclature. 341. [S.l.: s.n.] ISBN 9780121822422. ISSN 0076-6879. PMID 11582793. doi:10.1016/s0076-6879(01)41166-9
- ↑ «Cloning of the cDNA encoding the large subunit of human RNase HI, a homologue of the prokaryotic RNase HII». Proceedings of the National Academy of Sciences of the United States of America. 95. ISSN 0027-8424. PMC 23637
. PMID 9789007. doi:10.1073/pnas.95.22.12872
- ↑ «Yeast RNase H(35) is the counterpart of the mammalian RNase HI, and is evolutionarily related to prokaryotic RNase HII». FEBS Letters. 421. ISSN 0014-5793. PMID 9462832. doi:10.1016/s0014-5793(97)01528-7
- ↑ «RNase H2 of Saccharomyces cerevisiae is a complex of three proteins». Nucleic Acids Research. 32. ISSN 1362-4962. PMC 373335
. PMID 14734815. doi:10.1093/nar/gkh209
