Cladística
- "Métodos cladísticos" de análisis no es lo mismo que "escuela cladista" de clasificación.
La cladística (del griego κλάδος, klados: «rama») es una rama de la biología que define las relaciones evolutivas entre los organismos basándose en similitudes derivadas. Es la más importante de las sistemáticas filogenéticas que estudian las relaciones evolutivas entre los organismos. La cladística es un método de análisis riguroso que utiliza las "propiedades derivadas compartidas" (sinapomorfias: ver abajo) de los organismos que se están estudiando. El análisis cladístico forma la base de la mayoría de los sistemas modernos de clasificación biológica que buscan agrupar a los organismos por sus relaciones evolutivas. En contraste, la fenética agrupa los organismos basándose en su similitud global, mientras que los enfoques más tradicionales tienden a basarse en caracteres clave. Willi Hennig (1913-1976) es ampliamente considerado como el fundador de la cladística.
Esta clasificación será la más útil en campos de la biología como el estudio de la distribución de plantas (fitogeografía), las interacciones anfitrión-parásito y las interacciones planta-herbívoro, la biología de la polinización, la dispersión del fruto, o en contestar preguntas relacionadas con el origen de caracteres adaptativos (Nelson y Platnick, 1981;[1] Humphries y Parenti, 1986;[2] Brooks y McLennan, 1991;[3] Forey et al., 1992[4]). Debido a su marco teórico predictivo, una clasificación filogenética puede dirigir la búsqueda de genes, productos biológicos, agentes de biocontrol, y especies con potencial valor agrícola. La información filogenética también es útil para tomar decisiones relacionadas con la conservación. Finalmente, las clasificaciones filogenéticas proveen un marco teórico para el conocimiento biológico y una base para los estudios comparativos que enlazan todos los campos de la biología (Cracraft y Donoghue, 2004;[5] Judd et al. 2007: p. 32.).
Cladogramas
[editar]
Como resultado final del análisis cladístico se obtienen diagramas de relación en árbol llamados "cladogramas", para mostrar las distintas hipótesis sobre las relaciones. Un análisis cladístico puede basarse en tanta información como el investigador seleccione. La investigación sistemática moderna suele basarse en una gran variedad de información, incluyendo secuencias de ADN, ARN y proteínas (los famosos "datos moleculares"), datos bioquímicos y datos morfológicos.
En un cladograma todos los organismos se colocan en las hojas, y cada nodo interior es idealmente binario (con dos ramas). Los dos taxones de cada bifurcación se llaman taxones hermanos o grupos hermanos. Cada subárbol, independientemente del número de elementos que contenga, se llama clado. Todos los organismos de un grupo natural están contenidos en un clado que comparte un ancestro común (uno que no compartan con ningún otro organismo del diagrama). Cada clado se define en una serie de características que aparecen en sus miembros, pero no en las otras formas de las que ha divergido. Estas características identificadoras del clado se llaman sinapomorfias (caracteres compartidos derivados). Por ejemplo, las alas anteriores endurecidas (élitros) son una sinapomorfia de los escarabajos, mientras que la vernación circinada, o desenrollar los brotes nuevos, es una sinapomorfia de los helechos.
Definiciones
[editar]
- Apomorfía y plesiomorfía: un estado de carácter que esté presente en los dos grupos externos (los parientes más cercanos del grupo que no son parte del propio grupo) y en los ancestros se llama plesiomorfia (que significa "forma cercana", también llamado estado ancestral). Un estado de carácter evolutivamente novedoso, es decir, derivado de otro rasgo perteneciente a un taxón ancestral filogenéticamente próximo, se denomina apomorfia. Se utilizan los adjetivos plesiomórfico y apomórfico en lugar de "primitivo" y "avanzado" para evitar realizar juicios de valor sobre la evolución de los caracteres de estado, ya que ambos pueden ser ventajosos en distintas circunstancias.
- Sinapomorfía y simplesiomorfía: Una sinapomorfía es una apomorfía compartida por un grupo monofilético; una simplesiomorfía es una plesiomorfía compartida por un grupo parafilético.
- Se definen varios términos más para describir los cladogramas y las posiciones de los elementos dentro de ellos. Una especie o clado es basal de otro clado si tiene más caracteres plesiomórficos que el otro clado. Normalmente, un grupo basal es muy pobre en especies comparado con un grupo más derivado. No se requiere que haya un grupo basal. Por ejemplo, si se consideran las aves y los mamíferos juntos, ninguno es basal del otro: ambos tienen muchos caracteres derivados.
- Un clado o especie situado dentro de otro clado se puede describir como anidado dentro del otro clado.
Métodos cladísticos
[editar]Un análisis cladístico se aplica a un cierto conjunto de informaciones. Para organizar esta información se hace una distinción entre caracteres y estados de carácter. Si consideramos el color de las plumas, este puede ser azul en una especie, pero rojo en otra. Por tanto, las "plumas rojas" y las "plumas azules" son dos estados de carácter del carácter "color de las plumas".
En la época anterior al desarrollo de métodos computacionales automáticos, el investigador decidía qué estados de carácter estaban presentes antes del último ancestro común del grupo de especies (plesiomorfias) y cuáles estaban presentes en el último ancestro común (sinapomorfias). Normalmente, esto se hace considerando uno o más grupos externos (organismos que no se consideran parte del grupo en cuestión, pero que están relacionados con el grupo). Al caracterizar las divisiones cladísticas, solo las sinapomorfias son de utilidad. A continuación se dibujaban y evaluaban varios cladogramas posibles. Los clados debían tener tantas sinapomorfias como fuera posible. La esperanza es que un número bastante grande de verdaderas sinapomorfias será suficiente para superar a cualquier número de simplesiomorfias involuntarias (homoplasias), causadas por evolución convergente (es decir, caracteres que se parecen entre ellos por las condiciones ambientales o la funcionalidad, no por un ancestro común). Un ejemplo conocido de homoplasia por evolución convergente son las alas. Aunque las alas de las aves y los insectos pueden parecerse superficialmente y proporcionar la misma función, ambas evolucionaron independientemente. Si se puntuara como "POSITIVO" accidentalmente a un ave y un insecto para el carácter "presencia de alas", se introduciría una homoplasia en el conjunto de datos, lo que podría producir resultados erróneos.
Cuando surgen posibilidades equivalentes, se suele elegir una de ellas basándose en el principio de parsimonia: la disposición más compacta suele ser la mejor hipótesis sobre las relaciones. Otro enfoque, particularmente útil en la evolución molecular, es el de máxima verosimilitud, que selecciona el cladograma óptimo que tenga la mayor verosimilitud, basándose en un modelo de cambios de probabilidad específica.
Con el fin de obtener cladogramas de un modo lo más neutral posible, hoy en día gran parte del análisis se realiza por software: aparte del software para calcular los propios árboles, existe software estadístico sofisticado para proporcionar una base más objetiva.
La cladística ha tardado un tiempo en establecerse, y todavía hay gran debate sobre cómo aplicar las ideas de Hennig en el mundo real. Existe inquietud sobre el hecho de que la utilización de conjuntos de datos muy distintos (por ejemplo, características estructurales vs. características genéticas) puede producir árboles muy distintos. Sin embargo, en gran parte, la cladística ha demostrado su utilidad para resolver filogenias y se ha ganado un amplio respaldo.
La simplificación de la secuenciación de ADN y la sistemática computacional, que permite trabajar con grandes conjuntos de datos, los datos moleculares se utilizan cada vez más en la construcción de filogenias objetivas. De este modo, pueden distinguirse con mayor precisión las sinapomorfias de las homoplasias. Un método poderoso para reconstruir filogenias es el uso de marcadores retrotransposón genómicos, que están virtualmente libres de ambigüedad de acuerdo con el conocimiento actual (aunque esto es una suposición basada en estadísticas y puede no ser cierto en algún caso específico, aunque es improbable). Idealmente, se deberían combinar las filogenias morfológicas, moleculares y posiblemente otras (comportamentales, etc.): ninguno de los métodos es "superior", pero todos tienen distintas fuentes de error intrínsecas. Por ejemplo, la verdadera convergencia de caracteres es mucho más común en morfología que en las secuencias moleculares, pero las verdaderas reversiones de caracteres suelen ocurrir solo en estas últimas. La datación basada en información molecular es normalmente más precisa que la datación de fósiles, pero más cargada de errores (ver reloj molecular). Combinando y comparando se pueden eliminar muchos errores.
Clasificación cladística
[editar]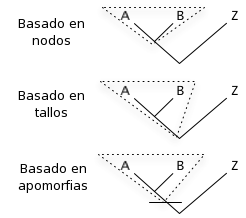
Basado en nodos: el ancestro común más reciente de A y B y todos sus descendientes.
Basado en tallos: todos los descendientes del más viejo ancestro común de A y B que no es ancestro de Z.
Basado en apomorfias: el ancestro común más reciente de A y B que posee una cierta apomorfia (carácter derivado), y todos sus descendientes.
La taxonomía cladística requiere que los taxones sean clados. En otras palabras, los cladistas argumentan que el sistema de clasificación debe reformarse para eliminar todo lo que no sean clados. En contraste, otros taxonomistas insisten en que los grupos reflejen las filogenias y hagan uso frecuente de las técnicas cladísticas, pero que se permitan grupos monofiléticos y parafiléticos como taxones.
Un grupo monofilético es un clado que comprende a una forma ancestral y todos sus descendientes, formando así un (y solo un) grupo evolutivo. Un grupo parafilético es similar, pero excluye a algunos de los descendientes que han sufrido cambios significativos. Por ejemplo, la clase tradicional Reptilia excluye a las aves, aunque estas evolucionaron a partir de un reptil ancestral. Igualmente, los invertebrados tradicionales son parafiléticos porque se excluyen los vertebrados, aunque los últimos evolucionaron a partir de un invertebrado.
Un grupo con miembros de líneas evolutivas separadas se llama polifilético. Por ejemplo, un grupo reconocido antiguamente, los paquidermos, resultó ser polifilético porque los elefantes y los rinocerontes surgieron separadamente de animales no paquidermos. Los taxonomistas evolutivos consideran que los grupos polifiléticos son errores en la clasificación, que ocurren porque se malinterpretó la convergencia o alguna otra homoplasia como una homología.
Siguiendo a Hennig, los cladistas argumentan que la parafilia es igual de dañina que la polifilia. La idea es que los grupos monofiléticos pueden definirse objetivamente, en términos de ancestros comunes o de la presencia de sinapomorfias. En cambio, los grupos parafiléticos y polifiléticos se definen por caracteres clave, y la decisión de qué caracteres son de importancia taxonómica es inherentemente subjetiva. Muchos argumentan que conducen a un pensamiento "gradístico", por el que los grados "bajos" avanzan hacia grados "avanzados", y que a su vez pueden conducir a una teleología. En los estudios evolutivos se suele evitar la teleología porque implica un plan que no puede demostrarse empíricamente.
Yendo más allá, algunos cladistas argumentan que los rangos de los grupos de especies son demasiado subjetivos para que representen ninguna información útil, y, por tanto, argumentan que deberían abandonarse. Por esta razón, se han alejado de la taxonomía linneana hacia una simple jerarquía de clados.
Otros sistemáticos evolutivos argumentan que todos los taxones son inherentemente subjetivos, aunque reflejen relaciones evolutivas, ya que los seres vivos forman un árbol esencialmente continuo. Toda línea divisoria es artificial y crea una sección monofilética por arriba y una sección parafilética por debajo. Los taxones parafiléticos son necesarios para clasificar secciones antiguas del árbol —por ejemplo, los primeros vertebrados que evolucionarían en la familia Hominidae no pueden situarse en ninguna otra familia monofilética—. También argumentan que los taxones parafiléticos proporcionan información sobre cambios significativos en la morfología de los organismos, en la ecología o en la historia de la vida —resumiendo, que tanto los taxones como los clados son nociones valiosas, pero con propósitos distintos—. Muchos usan el término monofilia en su sentido antiguo, en el que incluye a la parafilia, y usan el término alternativo holofilia para describir a los clados (monofilia en el sentido de Hennig).
Actualmente, está en desarrollo un código formal para la nomenclatura filogenética, el PhyloCode, para su uso en la taxonomía cladística. Se pretende que lo utilicen tanto los que quieren abandonar la taxonomía linneana como los que quieren usar taxones y clados al mismo tiempo.
Clasificación filogenética
[editar]

La clasificación filogenética es una clasificación científica de las especies basada únicamente en las relaciones de proximidad evolutiva entre las distintas especies, reconstruyendo la historia de su diversificación (filogénesis) desde el origen de la vida en la Tierra hasta la actualidad. Para construir este tipo de clasificación se recurre ahora generalmente al método cladístico, ideado por Willi Hennig y propuesto en su obra Grundzüge einer Theorie der phylogenetischen Systematik (Fundamentos de una teoría de la sistemática filogenética),[6] publicada en Alemania en 1950. Esta obra no obtuvo aceptación general en la comunidad hasta que Hennig la tradujera al inglés en 1966. Desde entonces la clasificación filogenética no ha cesado de reemplazar progresivamente a la clasificación tradicional (o linneana), iniciada por Carlos Linneo en 1735 en su libro Systema Naturae.
Varias ideas del análisis filogenético de las especies biológicas han sido incorporadas por la lingüística histórica en la clasificación filogenética de las familias de lenguas[7] y también por la crítica textual para la filiación de los diferentes manuscritos (testimonios) de una obra.[8]
Herramientas para el análisis filogenético
[editar]- Morfología (alternativa en animales y plantas).
- Secuencia de ADN, ARN y proteínas.
- Métodos probabilísticos:
Ejemplo
[editar]Se adjunta la clasificación filogenética del clado Aves: Las aves estarían categorizadas como una clase homónima en la taxonomía de Linneo. Pero en la taxonomía filogenética, las aves se ubican en el clado Theropoda (dinosaurios carnívoros bípedos).[9]
El establecimiento del origen dinosauriano del clado Aves ha tenido como consecuencia su clasificación filogenética con anidación sucesiva dentro de los siguientes clados:
| Rango del clado | Nombre del clado | Contenido del clado |
|---|---|---|
| (Mundo)/Imperio | Biota | Entidades biológicas, compuesto por biomoléculas |
| Subimperio | ¿Eunucleacuea? | |
| Sin rango | Deoxybiota/Eudeoxybiota | Entidades biológicas con ADN |
| Sin rango | Neodeoxybiota | |
| Dominio | Varidnaviria | Acelulares con rollo de gelatina |
| Sin rango | Cytotamorpha | Celuloides, protobiontes |
| Superdominio | Cytota | Organismos celulares |
| Dominio | Neomura/Arkaryota | Arqueas (incluyendo eucariotas) |
| Sin rango | Proteoarchaeota | Proteoarqueotas |
| Sin rango | Eukaryomorpha | Eucariomorfos, asgardarqueotas |
| Dominio | Eukaryota | Organismos eucariotas, protistas |
| Subdominio | Neokaryota | Grupo de animales, plantas y hongos |
| Sin rango | Opimoda | Protistas más emparentados a opistocontos que a corticatos |
| Sin rango | Podiata | Clado de Amorfeos y Crumalios |
| Sin rango | Amorphea | Protistas con triple gen fusionado |
| Sin rango | Obazoa | Protistas más cercanos a opistocontos que a las amebas verdaderas |
| Superreino[10] | Opisthokonta | Protistas que se desplazan por un flagelo posterior, Opistocontos |
| Sin rango | Holozoa | Opistocontos más emparentados a animales que a hongos |
| Sin rango | Filozoa | |
| Sin rango | Apoikozoa | Opistocontos coloniales |
| Reino | Animalia | Animales |
| Sin rango | Epitheliozoa | Animales con epitelio |
| Subreino | Eumetazoa | Animales verdaderos |
| Sin rango | ParaHoxozoa | Animales con Genes Hox |
| Infrareino | Bilateria | Animales bilaterales |
| Sin rango | Nephrozoa | Animales con sistema renal o similar, gusanos |
| Superfilo | Deuterostomia | Deuteróstomos |
| Filo | Chordata | Cordados, animales con notocorda |
| Subfilo | Vertebrata | Vertebrados |
| Infrafilo | Gnathostomata | Gnatóstomos, peces con mandíbula |
| Sin rango | Eugnathostomata | Peces no placodermos |
| Sin rango | Teleostomi | Grupo de osteíctios y acantodios |
| Sin rango | Euteleostomi | Vertebrados con lepidotricha, peces óseos y derivados |
| Clase | Sarcopterygii | Peces de aletas lobuladas |
| Sin rango | Rhipidistia | |
| Sin rango | Tetrapodomorpha | Peces más emparentados a los tetrápodos que a los peces pulmonados |
| Sin rango | Eotetrapodiformes | |
| Sin rango | Elpistostegalia | |
| Sin rango | Stegocephalia | Tetrápodos primitivos |
| Superclase | Tetrapoda | Tetrápodos modernos |
| Sin rango | Reptiliomorpha | Tetrápodos más emparentados a los amniotas que a los anfibios modernos |
| Sin rango | Amniota | Amniotas, reptiles tradicionales |
| Clase | Sauropsida | Amniotas más emparentados a los reptiles modernos que a los mamíferos |
| Sin rango | Eureptilia | Reptiles verdaderos |
| Sin rango | Romeriida | |
| Subclase | Diapsida | Reptiles diápsidos |
| Sin rango | Neodiapsida | Diápsidos avanzados |
| Sin rango | Sauria | Reptiles modernos |
| Sin rango | Archelosauria | Arquelosaurios, Grupo de tortugas y arcosaurios |
| Infraclase | Archosauromorpha | Animales más emparentados a los arcosaurios que a las tortugas e ictiosaurios |
| Sin rango | Crocopoda | |
| Sin rango | Archosauriformes | Arcosauriformes, Tecodontos |
| Sin rango | Eucrocopoda | |
| Sin rango | Crurotarsi | Crurotarsios, Grupo de arcosaurios y fitosaurios |
| Sin rango | Archosauria | Arcosaurios, Grupo de cocodrilianos y aves |
| Sin rango | Avemetatarsalia | Arcosaurios más emparentados a las aves que a los cocodrilianos |
| Sin rango | Ornithodira | Grupo de dinosaurios y pterosaurios |
| Sin rango | Dinosauromorpha | Animales más emparentados a los dinosaurios que a los pterosaurios |
| Sin rango | Dinosauriformes | |
| Sin rango | Dracohors | |
| Superorden | Dinosauria | Dinosaurios |
| Orden | Saurischia | Saurisquios, dinosaurios más emparentados a los saurópodos que a los ornitisquios. |
| Sin rango | Eusaurischia | Grupo de saurópodos y terópodos |
| Sin rango | Theropoda | Dinosaurios terópodos |
| Sin rango | Eutheropoda | |
| Sin rango | Averostra | |
| Sin rango | Tetanurae | |
| Sin rango | Orionides | |
| Sin rango | Tetanurae | |
| Sin rango | Avetheropoda | |
| Sin rango | Coelurosauria | |
| Sin rango | Tetanurae | |
| Sin rango | Tyrannoraptora | Grupo de Tyrannosaurus Rex y Aves |
| Sin rango | Maniraptoromorpha | |
| Sin rango | Maniraptoriformes | |
| Sin rango | Maniraptora | |
| Sin rango | Pennaraptora | |
| Sin rango | Paraves | |
| Sin rango | Eumaniraptora | |
| Sin rango | Avialae | Aves primitivas |
| Clase | Aves | Aves |
Aplicaciones
[editar]La cladística no asume ninguna teoría de la evolución particular, solo el conocimiento original de la descendencia con modificación. Por ello, los métodos cladísticos se pueden aplicar, y así se ha hecho recientemente, a sistemas no biológicos, como determinar las familias de lenguajes en la lingüística histórica y filiar manuscritos en la crítica textual, además de la clasificación artefactual en arqueología.
Véase también
[editar]- Escuelas de la Sistemática
- Taxonomía
- Análisis filogenético
- Filogenia
- Árbol filogenético
- Sistemática
- Taxonomía numérica
- Último ancestro común universal
- Willi Hennig
Referencias
[editar]- ↑ Nelson, G; Platnick, N. (1981). Systematics and biogeography. Nueva York: Columbia University Press.
- ↑ Humphries, C. J.; Parenti, L. R. (1986). Cladistic biogeography. Oxford: Clarendon.
- ↑ Brooks, D. R.; McLennan, D. A. (1991). Phylogeny, ecology and behavior. Chicago: University of Chicago Press.
- ↑ Forey, P. L.; Humphries, C. J.; Kitching, I. L.; Scotland, R. W.; Siebert, D. J.; Williams, D. M. (1992). Cladistics: a practical course in systematics. Oxford: Oxford University Press.
- ↑ Cracraft, J.; Donoghue, M. J. (2004). Assembling the tree of life. Oxford y Nueva York: Oxford University Press.
- ↑ Hennig. (1950) Grundzüge einer Theorie der phylogenetischen Systematik. Berlín: Deutscher Zentralverlag.
- ↑ Muñoz-Acebes, J. (2022) «Cladística e Historia de la Lengua Alemana: introducción al uso de PAUP». Revista de Humanidades Digitales, 7: 40-56
- ↑ Barbrook, A. C.; Howe, Ch. J.; Blake, N.; Robinson, P. (1998) «The phylogeny of The Canterbury Tales» Nature 394: 839.
- ↑ Livezey, Bradley C. & Zusi, Richard L. (2007). «Higher-order phylogeny of modern birds (Theropoda, Aves: Neornithes) based on comparative anatomy. II. Analysis and discussion.». Zoological Journal of the Linnean Society 149 (1): 1-95. Consultado el 29 de marzo de 2009.
- ↑ «El Superreino Okisthokonta».
Bibliografía
[editar]- de Queiroz, K; Gauthier, JA (1992) Phylogenetic taxonomy. Annual Review of Ecology and Systematics 23: pp. 449—480.
- de Luna, E., Guerrero, JA; Chew-Taracena, T. (2005). Sistemática biológica: avances y direcciones en la teoría y los métodos de la reconstrucción filogenética. Hidrobiológica 15 (3): pp. 351—370.
- Goloboff, PA. (1998) Principios básicos de cladística. Buenos Aires: Sociedad Argentina de Botánica.
- Hamdi, H; Nishio, H; Zielinski, R; Dugaiczyk, A (1999) Origin and phylogenetic distribution of “Alu” DNA repeats: irreversible events in the evolution of primates. Journal of Molecular Biology 289: pp. 861—871.
- Hennig, W. (1960) Elementos de una sistemática filogenética. Buenos Aires: EUDEBA.
- Kitching, IJ; Forey, PL; Humphries, CJ; Williams, DM (1998) Cladistics. (Segunda edición). Oxford: Oxford University Press.
- Muñoz-Acebes, J. (2022), Cladística e Historia de la Lengua Alemana: introducción al uso de PAUP, Revista de Humanidades Digitales, pp. 40-56.
- Patterson, C. (1982) Morphological characters and homology. En Joysey, KA; Friday, AE, eds. Problems in Phylogenetic Reconstruction. London: Academic Press.
- Shedlock, AM; Okada, N (2000) SINE insertions: Powerful tools for molecular systematics. Bioessays 22: pp. 148—160.
- Swofford, DL; Olsen, GJ; Waddell, PJ; Hillis, DM. (1996) «Phylogenetic inference». En Hillis, DM; Moritz, C; Mable, BK, ed. Molecular Systematics. Sunderland, MA: Sinauer Associates.
- Wiley EO. (1981) Phylogenetics: The Theory and Practice of Phylogenetic Systematics. Nueva York: Wiley Interscience.
Enlaces externos
[editar]Wikimedia Commons alberga una categoría multimedia sobre Cladística.
- La Sociedad Willi Hennig
- Tree of Life Web Project
- Filogenética.org: Plataforma de divulgación de la comunidad filogenética
- Controversias en sistemática filogenética Archivado el 8 de marzo de 2010 en Wayback Machine.
- Conceptos básicos en sistemática filogenética: los deuterostomados como ejemplo
- «El potencial de la evidencia fósil en la reconstrucción filogenética Archivado el 15 de febrero de 2009 en Wayback Machine.» Por Víctor M. Bravo Cuevas y Katia Adriana González Rodríguez